Analyse de suivie avec R
Pour faire l’analyse de suivie avec le Logiciel R nous allons utilisé deux packages R :
- survie pour le calcul d’analyses de survie
- survminer pour résumer et visualiser les résultats de l’analyse de survie
Commençons par l’installation des packages et téléchargeons les librairies dans le repertoire de travail:
install.packages(c("survival", "survminer"))
library("survival")
library("survminer")Nous utiliserons la base de données sur le cancer du poumon disponibles dans le dossier de survie. chargeons la base de données:
data("lung")
head(lung)Nous abtenons le taleau suivant
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0
6 12 1022 1 74 1 1 50 80 513 0- inst : code de l’établissement
- time : Temps de survie en jours
- statut : statut de censure 1=censuré, 2=mort
- âge : âge en années
- sexe : Masculin=1 Féminin=2
- ph.ecog : score de performance ECOG (0=bon 5=mort)
- ph.karno : score de performance de Karnofsky (mauvais=0-bon=100) évalué par un médecin
- pat.karno : score de performance de Karnofsky évalué par le patient
- repas.cal : Calories consommées aux repas
- wt.loss : Perte de poids au cours des six derniers mois
Calculer les courbes de survie : survfit()
La fonction survfit ( ) [dans le package de survie ] peut être utilisée pour calculer l’estimation de survie de Kaplan-Meier. Ses principaux arguments incluent :
- un objet de survie créé à l’aide de la fonction Surv ()
- et l’ensemble de données contenant les variables.
Nous allons calculer les proailités de suivie selon le sexe, tapez ceci :
fit <- survfit(Surv(time, status) ~ sex, data = lung)
print(fit)Par défaut, la fonction print() affiche un bref résumé des courbes de survie. Il imprime le nombre d’observations, le nombre d’événements, la survie médiane et les limites de confiance pour la médiane.
Call: survfit(formula = Surv(time, status) ~ sex, data = lung)
n events median 0.95LCL 0.95UCL
sex=1 138 112 270 212 310
sex=2 90 53 426 348 550Si vous voulez afficher un résumé plus complet des courbes de survie, tapez ceci :
# Summary of survival curves
summary(fit)
# Access to the sort summary table
summary(fit)$tableLa fonction survfit () renvoie une liste de variables, comprenant les composants suivants :
- n : nombre total de sujets dans chaque courbe.
- temps : les points temporels sur la courbe.
- n.risque : le nombre de sujets à risque au temps t
- n.événement : le nombre d’événements qui se sont produits à l’instant t.
- n.censor : le nombre de sujets censurés, qui sortent de l’ensemble de risques, sans événement, à l’instant t.
- inférieure, supérieure : limites de confiance inférieure et supérieure pour la courbe, respectivement.
- strates : indique la stratification de l’estimation de la courbe. Si strates n’est pas NULL, il y a plusieurs courbes dans le résultat. Les niveaux des strates (un facteur) sont les étiquettes des courbes.
Accès à la valeur retournée par survfit() se fait par:
d <- data.frame(time = fit$time,
n.risk = fit$n.risk,
n.event = fit$n.event,
n.censor = fit$n.censor,
surv = fit$surv,
upper = fit$upper,
lower = fit$lower
)
head(d) time n.risk n.event n.censor surv upper lower
1 11 138 3 0 0.9782609 1.0000000 0.9542301
2 12 135 1 0 0.9710145 0.9994124 0.9434235
3 13 134 2 0 0.9565217 0.9911586 0.9230952
4 15 132 1 0 0.9492754 0.9866017 0.9133612
5 26 131 1 0 0.9420290 0.9818365 0.9038355
6 30 130 1 0 0.9347826 0.9768989 0.8944820Visualisez les courbes de survie
Nous utiliserons la fonction ggsurvplot () [dans le package Survminer R] pour produire les courbes de survie des deux groupes de sujets.
Il est également possible d’afficher :
- les limites de confiance à 95 % de la fonction survivante en utilisant l’argument conf.int = TRUE .
- le nombre et/ou le pourcentage d’ individus à risque par temps en utilisant l’option risk.table . Les valeurs autorisées pour risk.table incluent :
- TRUE ou FALSE spécifiant s’il faut afficher ou non le tableau des risques. La valeur par défaut est FALSE.
- « absolu » ou « pourcentage » : pour indiquer le nombre absolu et le pourcentage de sujets à risque par temps, respectivement. Utilisez « abs_pct » pour afficher à la fois le nombre absolu et le pourcentage.
- la valeur p du test Log-Rank comparant les groupes en utilisant pval = TRUE .
- ligne horizontale/verticale à la survie médiane en utilisant l’argument surv.median.line . Les valeurs autorisées incluent l’une des valeurs c(« none », « hv », « h », « v »). v : vertical, h : horizontal.
# Change color, linetype by strata, risk.table color by strata
ggsurvplot(fit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE, # Add risk table
risk.table.col = "strata", # Change risk table color by groups
linetype = "strata", # Change line type by groups
surv.median.line = "hv", # Specify median survival
ggtheme = theme_bw(), # Change ggplot2 theme
palette = c("#E7B800", "#2E9FDF"))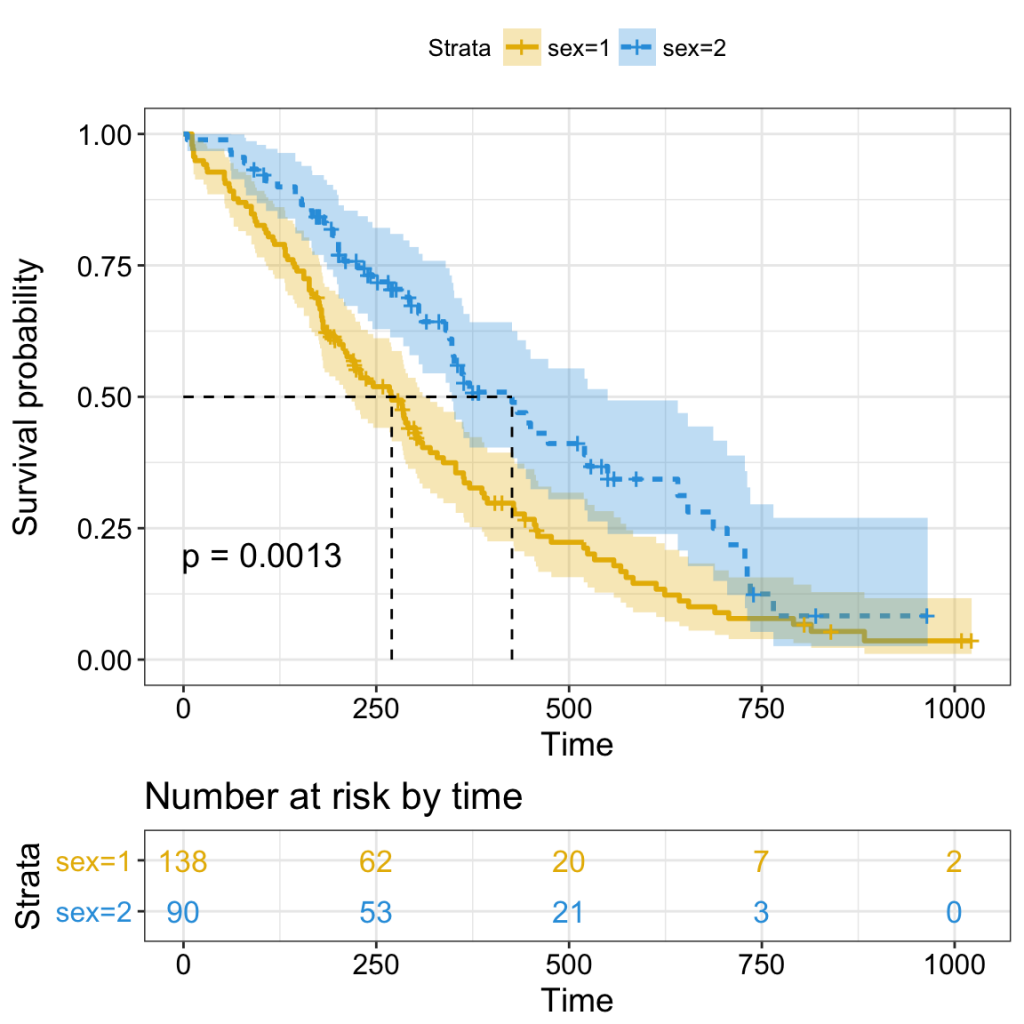
Le tracé peut être davantage personnalisé à l’aide des arguments suivants :
- conf.int.style = « step » pour changer le style des bandes d’intervalle de confiance.
- xlab pour changer l’étiquette de l’axe x.
- break.time.by = 200 casser l’axe x en intervalles de temps par 200.
- risk.table = « abs_pct » pour afficher à la fois le nombre absolu et le pourcentage d’individus à risque.
- risk.table.y.text.col = TRUE et risk.table.y.text = FALSE pour fournir des barres au lieu de noms dans les annotations textuelles de la légende du tableau des risques.
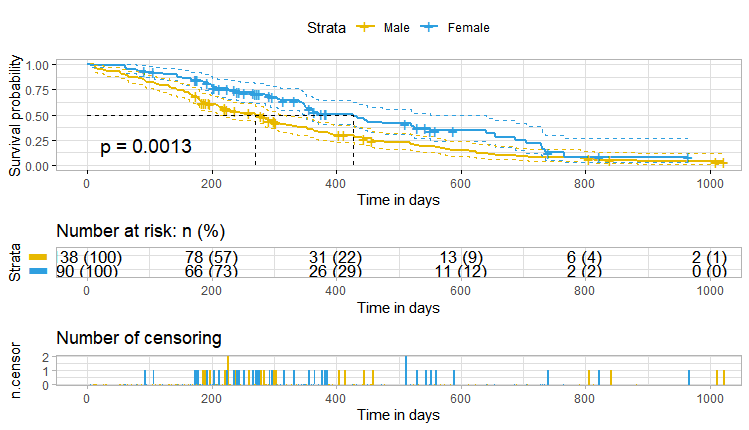
- ncensor.plot = TRUE pour tracer le nombre de sujets censurés au temps t. Comme l’a suggéré Marcin Kosinski , il s’agit d’un bon retour d’information supplémentaire sur les courbes de survie, afin que l’on puisse se rendre compte : à quoi ressemblent les courbes de survie, quel est le nombre de risques définis ET quelle est la cause pour laquelle le risque défini devient plus petit : est-ce causés par des événements ou par des événements censurés ?
- legend.labs pour modifier les étiquettes de légende.
ggsurvplot(
fit, # survfit object with calculated statistics.
pval = TRUE, # show p-value of log-rank test.
conf.int = TRUE, # show confidence intervals for
# point estimaes of survival curves.
conf.int.style = "step", # customize style of confidence intervals
xlab = "Time in days", # customize X axis label.
break.time.by = 200, # break X axis in time intervals by 200.
ggtheme = theme_light(), # customize plot and risk table with a theme.
risk.table = "abs_pct", # absolute number and percentage at risk.
risk.table.y.text.col = T,# colour risk table text annotations.
risk.table.y.text = FALSE,# show bars instead of names in text annotations
# in legend of risk table.
ncensor.plot = TRUE, # plot the number of censored subjects at time t
surv.median.line = "hv", # add the median survival pointer.
legend.labs =
c("Male", "Female"), # change legend labels.
palette =
c("#E7B800", "#2E9FDF") # custom color palettes.
)
Le diagramme de Kaplan-Meier peut être interprété comme suit :
L’axe horizontal (axe x) représente le temps en jours et l’axe vertical (axe y) montre la probabilité de survie ou la proportion de personnes survivantes. Les lignes représentent les courbes de survie des deux groupes. Une chute verticale dans les courbes indique un événement. La coche verticale sur les courbes signifie qu’un patient a été censuré à ce moment.
- Au temps zéro, la probabilité de survie est de 1,0 (ou 100 % des participants sont en vie).
- Au temps 250, la probabilité de survie est d’environ 0,55 (ou 55%) pour le sexe=1 et de 0,75 (ou 75%) pour le sexe=2.
- La survie médiane est d’environ 270 jours pour le sexe=1 et de 426 jours pour le sexe=2, suggérant une bonne survie pour le sexe=2 par rapport au sexe=1
Les temps de survie médians pour chaque groupe représentent le moment auquel la probabilité de survie, S(t), est de 0,5. Les temps de survie médians pour chaque groupe peuvent être obtenus à l’aide du code ci-dessous :
summary(fit)$table records n.max n.start events *rmean *se(rmean) median 0.95LCL 0.95UCL
sex=1 138 138 138 112 325.0663 22.59845 270 212 310
sex=2 90 90 90 53 458.2757 33.78530 426 348 550Le temps de survie médian pour le sexe = 1 (groupe masculin) est de 270 jours, contre 426 jours pour le sexe = 2 (féminin). Il semble y avoir un avantage de survie pour les femmes atteintes d’un cancer du poumon par rapport aux hommes. Cependant, pour évaluer si cette différence est statistiquement significative, il faut un test statistique formel, un sujet qui est abordé dans les sections suivantes.
L’analyse de survie est un ensemble d’approches statistiques pour l’analyse des données où la variable de résultat d’intérêt est le temps jusqu’à ce qu’un événement se produise.
Les données de survie sont généralement décrites et modélisées en termes de deux fonctions liées :
- la fonction de survie représentant la probabilité qu’un individu survive depuis le temps d’origine jusqu’à un certain temps au-delà du temps t. Il est généralement estimé par la méthode de Kaplan-Meier. Le test du logrank peut être utilisé pour tester les différences entre les courbes de survie des groupes, tels que les bras de traitement.
- La fonction de risque donne le potentiel instantané d’avoir un événement à un moment donné, compte tenu de la survie jusqu’à ce moment. Il est principalement utilisé comme outil de diagnostic ou pour spécifier un modèle mathématique pour l’analyse de la survie.
Dans cet article, nous montrons comment effectuer et visualiser des analyses de survie en utilisant la combinaison de deux packages R : survival (pour l’analyse) et survminer (pour la visualisation).
Cette analyse a été réalisée à l’aide du logiciel R (ver. 4.1.2).
Pour avoir plus d’explication ou pour une assistance dans une étude veuillez nous contacter au: info@labo-siber.com
